Arbres phylogénétiques, le making-of...

Un petit tuto rapide pour construire l'arbre phylogénétique, en supposant que vous avez réussi à obtenir d'une manière ou d'une autre la matrice des distances entre les différentes feuilles de cet arbre.
La première phase, et la plus compliquée, c'est de mettre cette matrice au format NEXUS, dont voici un exemple (tronqué, c'est juste pour comprendre) pour les distances-Eurovision entre les feuilles constituées par les pays européens :
#nexus
BEGIN Taxa;
DIMENSIONS ntax=39;
TAXLABELS
[1] 'Europe'
[2] 'Albania'
...
[39] 'Armenia'
;
END; [Taxa]
BEGIN Distances;
DIMENSIONS ntax=39;
FORMAT labels=left diagonal triangle=both;
MATRIX
[1] 'Europe' 0 8.03446523273329 6.94527480392245 6.08492529025624 6.42978267610967 7.61404519345663 5.99341744182733 6.90728067447675 6.92250335008947 6.7219201517126 6.68265626349283 7.07664803208412 6.71800408960876 6.32663542414134 6.01095491165921 7.08036573532752 7.29275054598743 6.54337752800494 6.95284871559852 6.66293755351196 7.30356798161008 8.31770464286873 7.08408148781478 6.89202437604511 6.01970448643453 6.74926896633998 7.00939219533334 6.99435862906671 6.86907639526596 6.67477576986673 6.60342813696038 6.59146178203287 7.63130671012508 6.75316689827817 6.55944477757074 7.28191704086225 6.89202437604511 6.43796306466572 7.17635004706431
...
[39] 'Armenia' 7.17635004706431 7.68114574786861 8.24621125123532 6.48074069840786 8.36660026534076 8.24621125123532 7.21110255092798 7.74596669241483 8.60232526704263 7.74596669241483 7.74596669241483 6.92820323027551 8.48528137423857 8 8.36660026534076 8.24621125123532 8.94427190999916 7.61577310586391 7.61577310586391 6.78232998312527 7.74596669241483 8.94427190999916 9.16515138991168 8.60232526704263 7.34846922834953 7.21110255092798 7.34846922834953 8.48528137423857 7.48331477354788 7.74596669241483 8.60232526704263 8.12403840463596 8.71779788708135 7.34846922834953 7.87400787401181 8.83176086632785 6.92820323027551 7.34846922834953 0
;
END; [Distances]
Alors certes, si vous l'avez dans un fichier Excel par exemple, vous pourrez vous arranger pour l'exporter en .CSV, puis jouer avec RECHERCHER/REMPLACER pour obtenir des espaces entre les différentes valeurs dans votre matrice... Mais il vaut mieux programmer un outil qui le fasse tout seul. Pour l'instant, je n'ai fait que des utilitaires très spécialisés au code affreusement crade, donc non distribuables en téléchargement direct sur internet...
Une fois ce pénible travail réalisé, passons au miracle Splitstree (4) dont vous aurez téléchargé auparavant la version adaptée à votre système d'exploitation (eh oui, c'est fait en Java, donc multiplateforme...) qui va trivialiser la fin de la construction.
Vous exécutez donc le programme, voilà à quoi ça ressemble :
Cliquez sur le 3° onglet, Source. Copiez-collez le code NEXUS dans cette fenêtre (ou utilisez le menu File, Open...), puis cliquez sur l'onglet Network. On vous avertit que l'entrée a changé et on vous suggère de prendre en compte les modifications (Input has change, execute it?), ce que vous acceptez sans frémir par un Yes assuré. Et là un truc affreux apparaît, un réseau phylogénétique.  Affreux parce que pas compréhensible par un oeil non exercé (en fait, le truc permet de visualiser plus d'informations qu'un arbre phylogénétique, mais bon, ne nous étendons pas là-dessus, et tentons plutôt d'obtenir un arbre classique...). Dans le menu Trees, choisissez donc BioNJ si vous êtes chauvin, NJ si vous voulez rédiger l'article Wikipedia qui explique la méthode, et UPGMA si vous vous moquez d'obtenir un mauvais arbre mais que vous tenez à savoir comment il a été construit. Disons que vous prenez BioNJ, cliquez sur le bouton Apply dans la fenêtre qui apparaît.
Affreux parce que pas compréhensible par un oeil non exercé (en fait, le truc permet de visualiser plus d'informations qu'un arbre phylogénétique, mais bon, ne nous étendons pas là-dessus, et tentons plutôt d'obtenir un arbre classique...). Dans le menu Trees, choisissez donc BioNJ si vous êtes chauvin, NJ si vous voulez rédiger l'article Wikipedia qui explique la méthode, et UPGMA si vous vous moquez d'obtenir un mauvais arbre mais que vous tenez à savoir comment il a été construit. Disons que vous prenez BioNJ, cliquez sur le bouton Apply dans la fenêtre qui apparaît.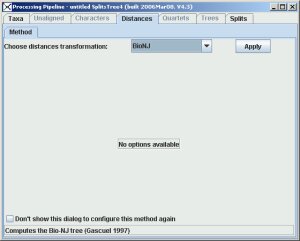

Vous obtenez alors (après Apply et fermeture de la fenêtre) le joli arbre suivant :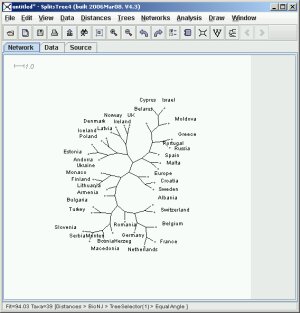
Et voilà, il ne "reste" plus qu'à interpréter le résultat, et en faire profiter les internautes (en citant www.splitstree.org) ou la communauté scientifique (en citant D. H. Huson and D. Bryant, Application of Phylogenetic Networks in Evolutionary Studies, Mol. Biol. Evol., 23(2):254-267, 2006).
D'autre part, Splitstree recèle d'astuces diverses, donc la lecture du manuel d'utilisation sera profitable à tout anglophone qui souhaite les découvrir.








3 commentaires:
Bonjour,
Savez vous s'il est possible de réaliser des arbres phylogénétiques à partir du vocabulaire, du style, ou encore des expressions de certains romans ou autres textes ? Etablir ainsi une sorte de filiation des oeuvres écrites.
Il me semble que cela a peut être été déjà fait ?
Il faudra que je regarde ça plus en détail, tout est une histoire de distances... Après on peut choisir n'importe quelle méthode de reconstruction de l'arbre à partir de la matrice des distances...
En allant visiter les pages web de Vitanyi et Cilibrasi, vous tomberez sur des trucs intéressants, sur le calcul de distances entre oeuvres musicales comme l'article de Jean-Paul Delahaye dans Pour la Science : Classer musiques, langues,
images, textes et génomes - http://homepages.cwi.nl/~paulv/papers/SimilarityV3.pdf.
Et en googlant distance de Jaccard, j'ai trouvé ça : http://revel.unice.fr/corpus/document.html?id=30.
Pas mal de trucs à creuser, et à implémenter, donc :).
Merci de votre réponse.
Si j'arrive à produire qqchose je vous informerez...
Enregistrer un commentaire